Программа ARGO_CUDA
Ссылка на программу
https://gitea.sysbio.ru/program_providing_fund/ArgoCUDA
Модуль «ARGO_CUDA» предназначен для выявления в последовательностях пиков ChIP-экспериментов значимых олигонуклеотидов. Осуществляет оценку сходства выявленных с помощью программы ArgoCUDA IUPAC мотивов с весовыми матрицами, хранящимися в БД Jaspar и Hocomoco. Модуль включает в себя следующие компоненты:
-
Оценка сходства IUPAC мотивов с весовыми матрицами осуществляется с помощью системы Tomtom (https://meme-suite.org/meme/tools/tomtom).
-
Результаты работы системы Tomtom анализируются программой MotifAnnotator_Base.
Описание входных данных
На вход конвейера подается файл «query_motifs» с IUPAC мотивами в fasta – формате и БД Hocomoco и Jaspar. Пример файлов «query_motifs» с IUPAC мотивами в fasta – формате (фрагмент):
>TRTWKACH
TRTWKACH
>RTTKACHY
RTTKACHY
>TMAAYANS
TMAAYANS
БД Hocomoco «HOCOMOCOv11_full_annotation_HUMAN_MOUSE.tab» и Jaspar «Jaspar.tab».
Описание выходных данных
-
Результат работы Tomtom – файл «tomtom.tsv». Пример результата в прилагаемых файлах «FoxA2_major_30_Hocomoco_0_01.tsv» (рисунок 1) и «FoxA2_major_30_Jaspar_redundant_2022_vertebrates_0_01.tsv» (рисунок 2).
-
Результат работы MotifAnnotator_Base – файл «result.out» (рисунок 3).
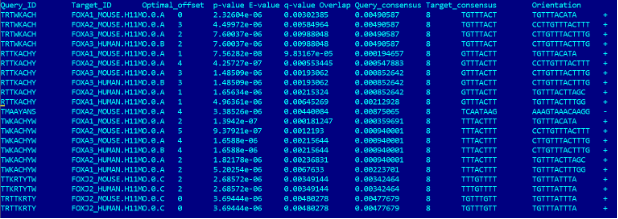
Рисунок 1 - Пример выходного файла «FoxA2_major_30_Hocomoco_0_01.tsv»
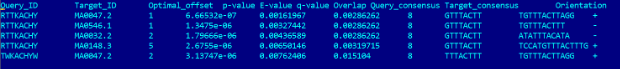
Рисунок 2 - Пример выходного файла «FoxA2_major_30_Jaspar_redundant_2022_vertebrates_0_01.tsv»
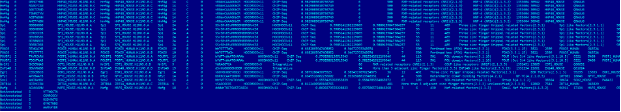
Рисунок 3 - Пример выходного файла «result.out».