Программный модуль “YPApred”
Программный модуль “YPApred”
Модуль предназначен для предсказания уровня представленности белков в клетках Saccharomyces cerevisiae S288C по нуклеотидной последовательности регуляторной области в размере 1000 пар оснований до старт-кодона гена и по аминокислотной последовательности кодируемого белка. Предсказание происходит в два основных этапа: получение векторных представлений последовательностей, предсказание уровня представленности белков по заданным векторным представлениям. Подробная схема работы представлена на Рис.1.
-
В начале поданные на вход последовательности векторизуются с помощью моделей ESM-2 для аминокислотной последовательностей и GENA-LM для нуклеотидных последовательностей. Векторизованные последовательности подаются на вход обученной регрессионной “головы” - обученной нейронной сети для предсказания количества белка на основе их векторизованных последовательностей. Для векторизации последовательности белка используется модель esm2_t33_650M_UR50D из семейства белковых языковых моделей ESM2, обученных на последовательностях белков из UniProt. Для векторизации нуклеотидной последовательности используется модель gena-lm-bert-base-yeast, обученная на геномных последовательностях пекарских дрожжей, из семейства моделей GENA-LM. При векторизации веса моделей остаются “замороженными”. Значения векторов получаются усреднением значений по всем токенам в одной оси после удаления специальных токенов CLS и SEP. Усреднение по токенам необходимо для получения векторов фиксированной длины для всех последовательностей. Все вектора получены в режиме half-precision.
-
Полученные векторные представления объединяются и подаются на вход регрессионного слоя. Регрессионный слой является полносвязной нейронной сетью. При обучении использовалась функция активации ReLU для каждого слоя. Количество нейронов во входном слое - 2048, выходного слоя - 1 (значение количества белка в клетке). Размер первого скрытого слоя 256 нейронов, размер второго скрытого слоя - 32 нейрона. Оптимизатором являлся Adam с коэффициентом скорости обучения равным 0.0001. Число эпох при обучении было 20. Функция ошибки – MSE. Данные делились на обучающие и тестовые выборки в соотношении 80 и 20% соответственно. Для реализации нейронной сети использовался пакет PyTorch.
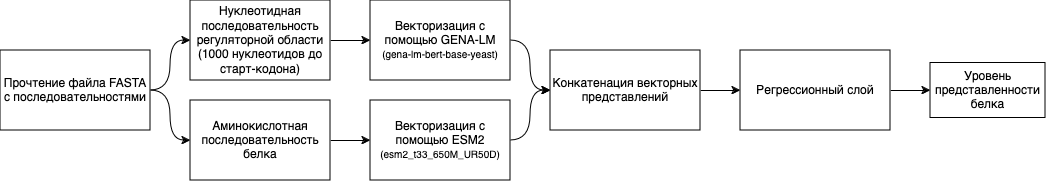
Рис. 1. Блок-схема программного модуля YPApred.
Описание входных данных.
На вход метода подается нуклеотидная последовательность регуляторной области и аминокислотная последовательность в файле формате FASTA.
Описание выходных данных
Логарифмирование по основанию 10 количество данного белка в клетке Saccharomyces cerevisiae S288C.