Дизайнер пептидных ингибиторов апоптоза
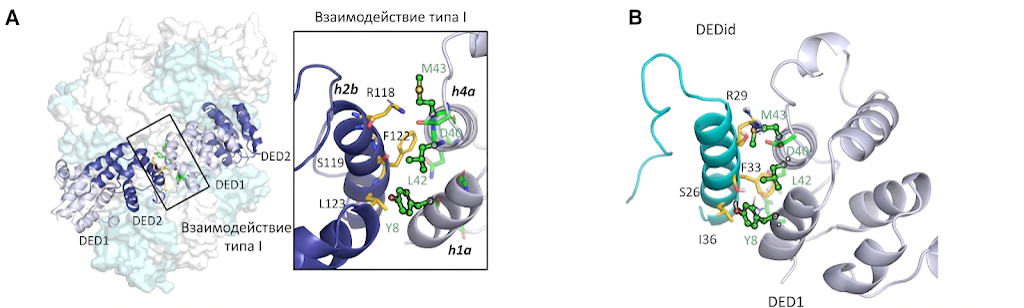
ВВЕДЕНИЕ. Изучение молекулярных механизмов программируемой клеточной гибели (апоптоза) представляет собой одну из ключевых задач современной молекулярной и клеточной биологии. Такие исследования важны для понимания патогенеза онкологических, аутоиммунных и нейродегенеративных заболеваний, а также для поиска новых терапевтических мишеней. Критическую актуальность в этих исследованиях приобретает анализ белок-белковых взаимодействий в сигнальном комплексе, индуцирующем смерть (DISC — Death-Inducing Signaling Complex), который является ключевой платформой для активации каспазы-8 и запуска внешнего пути апоптоза. Сборка DISC опосредована взаимодействиями доменов смерти (DED — Death Effector Domain), которые формируют филаментные структуры. Белки FADD, прокаспаза-8, прокаспаза-10 и c-FLIP образуют центральное ядро DISC посредством трёх типов асимметричных взаимодействий между соседними DED-доменами. Взаимодействия типа I опосредованы контактами между интерфейсами Ia (спирали h1a/h4a домена DED1) и Ib (спираль h2b домена DED2), при этом ключевую роль играет консервативный гидрофобный FL-мотив (F122/L123). Важной задачей для регулирования апоптоза является разработка молекулярных инструментов, способных селективно блокировать DED-взаимодействия. Для решения этой задачи был разработан информационно-программный модуль (ИПМ) рационального дизайна пептидов на основе RosettaScript. ИПМ использует структурную суперпозицию, белок-белковый докинг и энергетическую оптимизацию для моделирования пептидов-миметиков, имитирующих ключевые структурные элементы DED-интерфейсов. Разработка пептида DEDid, блокирующего взаимодействий типа I между доменами белков с DED доменами, участвующих в сборке сигнального комплекса DISC и регуляции апоптоза С помощью разработанного ИПМ был спроектирован пептид DEDid, имитирующий спираль h2b домена DED2 прокаспазы-8, содержащую FL-мотив. В качестве исходных данных использовались: (1) крио-ЭМ структура DED-филамента прокаспазы-8 (PDB: 5L08), (2) ЯМР-структура бычьего панкреатического полипептида (PDB: 1BBA) в качестве стабильного α-спирального скаффолда. Протокол моделирования включал следующие этапы:
- Идентификация ключевых остатков интерфейса типа I. На основе крио-ЭМ структуры были определены остатки, формирующие гидрофобный кластер: L42, Y8, M43 (интерфейс Ia DED1) и F122, L123 (FL-мотив интерфейса Ib DED2), а также остатки, образующие водородные связи: S115, R118, S119.
- Конструирование последовательности DEDid. В скаффолдную последовательность BPP были введены замены для имитации ключевых остатков h2b: R33F/R34L (имитация FL-мотива), A26S, A29R, A30S (имитация гидрофильных остатков S115, R118, S119). N-концевая последовательность R8 добавлена для обеспечения клеточной проницаемости.
- Молекулярное моделирование комплекса. Начальная структура комплекса прокаспаза-8-DED1/DEDid была получена путём структурной суперпозиции Cα-атомов спиральной области DEDid (остатки S26, R29, S30, L34, F33, N37) на Cα-атомы области h2b каспазы-8 (остатки S115, R118, S119, F122, L123, E126). Комплекс был оптимизирован с использованием высокоразрешающего протокола белок-белкового докинга Rosetta с последующей релаксацией.
- Отбор референсной структуры. Из ансамбля 100 конформаций отбирались 10% структур с наименьшей энергией связывания. Конформационная кластеризация проводилась с помощью программы Calibur. Поза связывания, соответствующая центру наибольшего кластера, была выбрана в качестве референсной модели. Результаты моделирования: Итоговая последовательность пептида DEDid (46 а.о.): Ac-RRRRRRRRAPLEPEYPGDNATPEQMSQYRSELFLYINML-NH2. Согласно предсказанной структуре комплекса, DEDid формирует гидрофобный кластер с остатками L42, Y8 и M43 прокаспазы-8-DED1 посредством остатков F33, L34 и I36 (рисунок Ж.41A,B). Остаток R29 DEDid образует солевой мостик с D40 прокаспазы-8-DED1, а N37 формирует водородные связи с Q46 и карбонильным кислородом остова спирали h1a. Аналогичным образом была построена модель комплекса DEDid/FADD-DED. Анализ показал, что интерфейс Ia FADD-DED содержит более выраженный гидрофобный карман (остатки L5, V6, H9, L43, L50), что может обеспечивать усиленное связывание с FL-мотивом DEDid. Дополнительно были спроектированы контрольные пептиды со скремблированной последовательностью (ctr1-3) и мутантный пептид ctr4 с заменами в FL-мотиве (F33D/L34S) для экспериментальной валидации специфичности связывания. Экспериментальная валидация: Предсказания модуля были подтверждены методом изотермической титрационной калориметрии (ITC): DEDid связывается с прокаспазой-8-C360A-FL (Kd ≈ 518 мкМ), тогда как контрольные пептиды и мутант прокаспазы-8 по сайту связывания (L42S/Y8S) не демонстрируют связывания (таблица Ж.8).
ЗАКЛЮЧЕНИЕ Таким образом, разработанный ИПМ позволяет осуществлять рациональный дизайн пептидов-ингибиторов DED-взаимодействий на основе структурных данных. Пептид DEDid, спроектированный с помощью данного модуля, продемонстрировал способность блокировать сборку DED-филаментов, активацию каспаз и CD95L-индуцированный апоптоз в различных клеточных линиях (HeLa-CD95, MV4-11, первичные T-клетки), что открывает перспективы для разработки терапевтических стратегий, направленных на модуляцию путей клеточной гибели.