Модуль для предсказания влияния мутаций на стабильность белков - AIProtMutEnergy.
Программный модуль (Рис.1) предназначен для предсказания влияния мутаций на стабильность пространственной структуры белка, предсказанной с помощью метода искусственного интеллекта ESM3.
Модуль начинает работу с обработки входных данных и с инициализации пакета молекулярного моделирования PyRosetta с начальными параметрами: -ex1, -ex2, -flip_HNQ, -relax:cartesian, -relax:default_repeats 5, -nstruct 100, -optimization:default_max_cycles 100. Параметры -ex1 и -ex2 позволяют расширить пространство разрешенных конформационных изменений для углов \(ꭓ1\) и \(ꭓ2\) боковых радикалов, улучшая точность моделирования, -flip_HNQ позволяет поворачивать водороды амидной цепи для гистидина, аспарагина и глутамина, что обеспечивает правильное положение водородных связей, -relax: cartesian и -relax:default_repeats 5 повторяют 5 раз каждый процесс минимизации структуры в декартовых координатах, -nstruct 100 каждый процесс минимизации или внесении мутации создает ансамбль из 100 структур с разными конформаций, откуда выбирается структура с минимальной энергией.
Далее для последовательности дикого типа белка предсказывается 10 структур в ESM3 с разными заданными значениями параметра seed от 1 до 10. Из предсказанных структур выбирается структура с минимальным предсказанным средним значением ошибки pLDDT. Предсказанная структура имеет только основную цепь. Предсказанная структура дополняется боковой цепью и минимизируется с помощью алгоритма FastRelax со значением баростата 1 атм. и протоколом изменения конформации MonteCarlo , где в качестве оценочной функции энергии структуры использовалась функция ref2015, которая была параметризована так, чтобы воспроизводить термодинамически наблюдаемые и структурно-основанные свойства белков. Далее вносятся в минимизированную структуру вносятся мутации с помощью модифицированного алгоритма CartesianDDG. Далее изменение свободной энергии мутантных белков вычисляется по формуле:
$$ddG = dGmut - dGwt,$$
где \(ddG\) - изменение свободной энергии в REU (Rosetta Energy Units), \(dGmut\)- свободная энергия мутанта, \(dGwt\) - свободная энергия дикого типа. Далее \(ddG\) были переведены в ккал/моль.
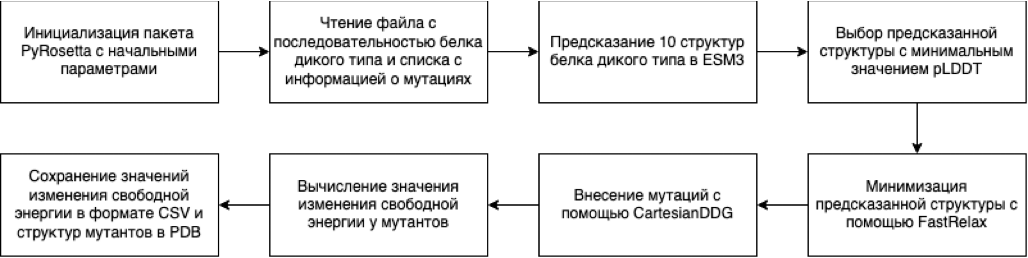
Рис. 1. Блок-схема работы программного конвейера.
Описание входных данных:
На вход конвейера подается последовательность белка дикого типа в формате FASTA, и список мутаций, где каждая мутация записывается в отдельной строке в следующем виде: A100B, где аминокислота A на 100 позиции в последовательности заменяется на аминокислоту B.
Описание выходных данных:
Предсказанная структура белка дикого типа, предсказанные структуры мутантных вариантов белка, изменение значения свободной энергии ddG и энергетических терм из оценочной функции ref2015 для каждой мутации.
Компоненты функции оценки энергии ref2015:
- fa_atr (Lennard-Jones attractive) - описывает силы Ван-дер-Ваальса притяжения между атомами;
- fa_rep (Lennard-Jones repulsive) - отвечает за силы отталкивания между атомами при их чрезмерном сближении;
- fa_sol (Lazaridis-Karplus solvation energy) - учитывает эффекты сольватации, то есть взаимодействия белка с водным окружением;
- fa_intra_rep (internal repulsive energy) - описывает внутримолекулярные силы отталкивания;
- fa_elec (coulombic electrostatic potential) - отражает электростатические взаимодействия между заряженными группами;
- pro_close и hbond_sc - специфические термы для описания взаимодействий с участием пролина и водородных связей боковых цепей соответственно;
- rama_prepro - оценивает конформационные предпочтения основной цепи на основе карт Рамачандрана;
- omega - характеризует торсионные напряжения пептидной связи;
- dslf_fa13 - описывает дисульфидные связи между остатками цистеина.