Конвейер «OrthoDom» для поиска последовательностей семейств многодоменных белков на основе ортологии и анализа доменов
Конвейер OrthoDom позволяет выполнять поиск последовательностей семейств многодоменных белков на основе ортологии и анализа доменов среди аминокислотных последовательностей, кодируемых в геномах исследуемых организмов.
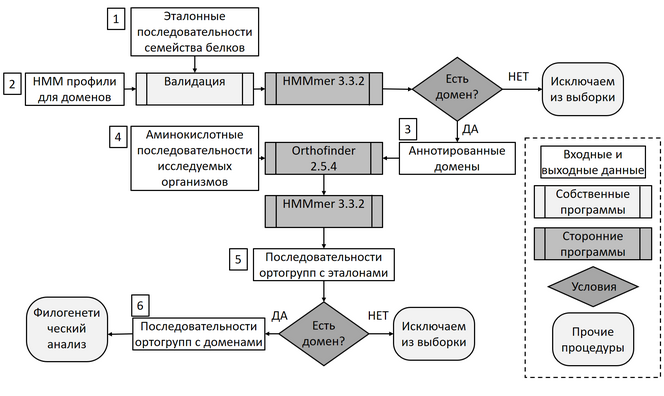
Рисунок 1 Блок-схема вычислительного конвейера OrthoDom. Обозначения блоков показаны в пунктирном прямоугольнике справа: прозрачными прямоугольниками показаны входные и выходные данные, серыми – собственные и сторонние программы, ромбами – условные операторы, скругленными прямоугольниками – другие процедуры с данными. Для основных входных и выходных данных в прямоугольных рамках приведены цифровые маркеры (см. текст).
В качестве входных данных (маркер 1 на рис. 1) используются последовательности исследуемого семейства многодоменных белковых последовательностей с качественной аннотацией (как правило, выявленных и аннотированных у модельных организмов). Для эталонных последовательностей указываются списки функциональных доменов, которые они включают. Для этих доменов из базы данных Pfam 33.1 извлекаются соответствующие профили HMM (маркер 2). Далее с помощью программы hmmsearch пакета HMMer 3.3.2, проводится валидация эталонных белков по наличию указанных доменов (маркер 3), так как для некоторых из них могут быть фрагментированы или отсутствовать. Еще один набор входных данных – аминокислотные последовательности (протеомы) исследуемых организмов (как правило, немодельных), в которых требуется определить ортологов эталонных белков (маркер 4). Ортологические группы для аминокислотных последовательностей эталонных белков и белков исследуемых организмов определялись программой OrthoFinder v. 2.5.4. Выявленные с помощью нее ортологические группы (маркер 5), включающие эталонные последовательности, дополнительно проверялись на наличие доменов. Выявленные таким образом последовательности ортологов эталонных белков (маркер 6) обрабатывались далее для реконструкции филогении программой IQ-TREE.
Описание входных данных
-
Эталонные последовательности белковых семейств. Последовательности могут принадлежать различным организмам. Последовательности одного организма должны находиться в отдельном файле в формате FASTA. Файлы эталонных последовательностей должны быть в отдельной папке.
-
Список профилей Pfam для доменов, которые должны быть представлены в структурах искомых белков. Профили должны быть представлены в файле формата HMMer.
-
Последовательности белков из исследуемых геномов. Для каждого генома должен быть сформирован файл в FASTA формате с аминокислотными последовательностями, которые он кодирует.
Описание выходных данных
Выходными файлами являются файлы, которые генерируются программой Orthofinder. Они располагаются в папке OrthoFinder/Results после выполнения конвейера.
/Comparative_Genomics_Statistics - папка с данными по статистике геномов
Orthogroups_SpeciesOverlaps.tsv - список ортогрупп ,представленных в различных геномах
Statistics_Overall.tsv – общая статистика по представленности ортогрупп в геномах
Statistics_PerSpecies.tsv – статистика представленности ортогрупп в отдельных
/Gene_Trees – папка с фафлами филогенетических деревьев для каждой ортогруппы в формате nph.
/Orthogroup_Sequences - папка с файлами последовательностей для каждой ортогруппы в FASTA формате
/Orthogroups – папка со списком генов в ортогруппах
Orthogroups.GeneCount.tsv – файл с данными по количеству генов в ортогруппах по геномам
Orthogroups.tsv, Orthogroups.txt – списки генов в ортогруппах в двух форматах
Orthogroups_SingleCopyOrthologues.txt – списки ортогрупп, в которых от каждого организма представлена только одна копия гена
Orthogroups_UnassignedGenes.tsv – списки генов, которые не вошли ни в одну ортогруппу.