Регуляторная сеть взаимодействия длинных некодирующих РНК с микроРНК кукурузы
ВВЕДЕНИЕ. Между длинными некодирующими РНК (днРНК) и микроРНК (миРНК) существуют сложные взаимодействия, значимые для регуляции роста, развития растений и ответов на стрессовые факторы. днРНК могут действовать как молекулярные “губки”, связываясь с миРНК и предотвращая их взаимодействие с целевыми мРНК. Анализ взаимодействия между днРНК и миРНК даёт ценную информацию о механизмах посттранскрипционной регуляции экспрессии генов, имеющих как фундаментальное значение для реконструкции механизмов, контролирующих фенотипические признаки растений, так и для практических приложений в области селекции сельскохозяйственных растений, что и определяет актуальность разработки описываемого программного модуля. Модуль предназначен для выявления потенциальных взаимодействий между днРНК и миРНК на основе анализа дифференциальной экспрессии и методов машинного обучения. Конвейер состоит из трех блоков: (1) сборка транскриптомов из данных RNA-seq с использованием программ STAR и StringTie, выявление дифференциально экспрессирующихся днРНК (2) предсказание взаимодействий между днРНК и миРНК с пониженной экспрессией у мутантов посредством алгоритма машинного обучения PmliPred, (3) верификация предсказанных пар с использованием программы IntaRNA для анализа структурной комплементарности и построение сетей конкурирующих эндогенных РНК (ceRNA). Для запуска анализа необходимы данные RNA-seq, референсный геном и информация о последовательностях миРНК. Результаты работы конвейера: выявленные днРНК, сети взаимодействия днРНК-микроРНК с оценкой вероятностей взаимодействия, а также визуализация этих сетей, демонстрирующаяпотенциальные функции днРНК как молекулярных «губок» для миРНК. Модуль обеспечивает углубленное понимание механизмов посттранскрипционной регуляции в растениях с участием днРНК, выявление регуляторных днРНК, перспективных для селекции, направленной на повышение стрессоустойчивости и продуктивности сельскохозяйственных культур. ОПИСАНИЕ ВХОДНЫХ ДАННЫХ. На вход модуля подаются данные RNA-seq в формате FASTQ, указывается путь доступа к референсному геному в формате FASTA с аннотацией, а также подаются последовательности зрелых микроРНК из базы miRBase. ОПИСАНИЕ РАБОТЫ МОДУЛЯ И ГЕНЕРИРУЕМЫХ ВЫХОДНЫХ ДАННЫХ. Анализ взаимодействий между днРНК и миРНК (рисунок Ж.36) включает три основных этапа. 1. Сборка и аннотация данных RNA-seq с использованием программы Trimmomatic для удаления адаптеров и некачественных прочтений, выравнивани\ прочтений на референсный геном с помощью программы STAR, сборка транскриптомов с использованием программы StringTie, выявление днРНК программой ICAnnoLncRNA и расчет уровней экспрессии через Kallisto. Оценка качества собранных транскриптомов проводится с помощью BUSCO. 2. Выявление дифференциально экспрессирующихся днРНК осуществляется посредством анализа различий в уровнях экспрессии с использованием пакетов DESeq2 и edgeR, отбора транскриптов с достоверными изменениями экспрессии (p-value < 0.05) и построения тепловых карт для визуализации паттернов экспрессии в различных биологических повторностях. 3. Предсказание и верификация взаимодействий включает (а) применение алгоритма машинного обучения PmliPred для идентификации пар днРНК-микроРНК (с расчетом доверительной вероятности (PC) взаимодействия); (б) отбор пар с PC ≥ 0.5 в тестовой группе; (в) верификацию предсказанных взаимодействий программой IntaRNA путем анализа структурной комплементарности и (г) отбор пар днРНК-микроРНК, удовлетворяющих следующим условиям: длина области связывания более 16 нуклеотидов и наличие менее 4-х неспаренных оснований.
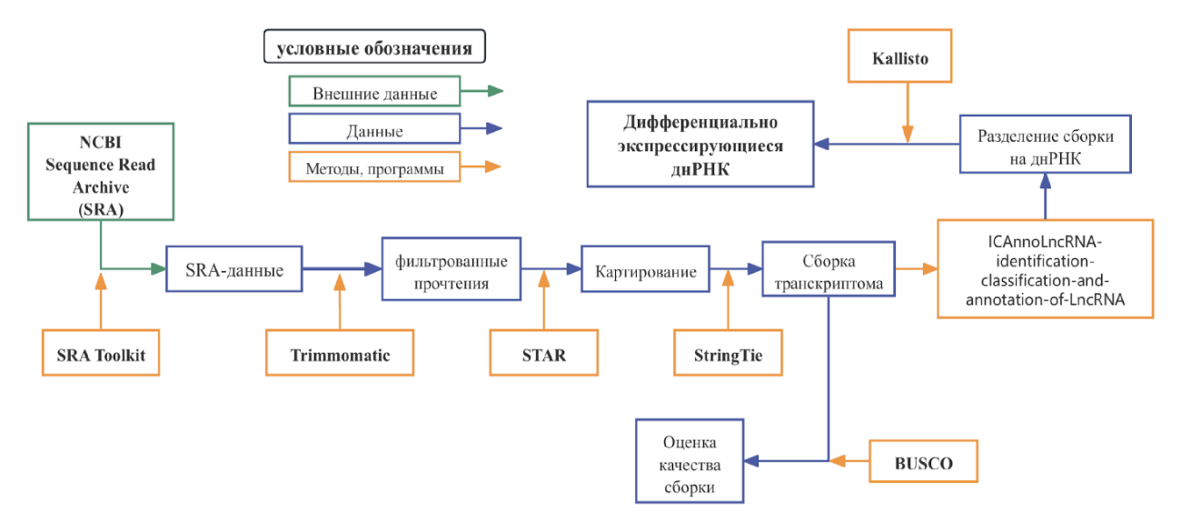
Рисунок Ж.36 – Схема анализа взаимодействий между днРНК и миРНК, реализованная в программном модуле
Перечень программного обеспечения и баз данных, необходимых для работы ипм, включая блок-схему: Trimmomatic, STAR, StringTie, BUSCO, Kallisto, DESeq2, edgeR, ICAnnoLncRNA, PmliPred, IntaRNA, Cytoscape, NCBI SRA, Ensembl Plants, miRBase. РЕЗУЛЬТАТЫ РАБОТЫ МОДУЛЯ: (а) таблицы дифференциально экспрессирующихся днРНК с указанием их идентификаторов и уровней экспрессии, (б) предсказанные взаимодействия днРНК-микроРНК с вероятностными оценками (PC-значения), (в) структурные характеристики взаимодействий (длины областей связывания, минимальные свободные энергии), (г) сети конкурирующих эндогенных РНК (ceRNA-сети), визуализированные в программе Cytoscape, демонстрирующие потенциальные функции днРНК как молекулярных «губок» и способность отдельных днРНК связывать несколько миРНК одновременно, а также (д) списки миРНК-партнеров для каждой днРНК с известными функциями в регуляции роста, развития и ответов на стресс растений. ДЕМОНСТРАЦИОННЫЙ ПРИМЕР. На основе данных RNA-seq для побега кукурузы выявлено 10 дифференциально экспрессирующихся днРНК, из них 9 повышают экспрессию у мутантов по отношению к контрольным растениям. Для соцветия выявлено 34 дифференциально экспрессирующихся днРНК, из них 20 повышают экспрессию у мутантов. Комплексный анализ выявил потенциальные взаимодействия между днРНК и миРНК в мутантной линии кукурузы. Предсказание с использованием PmliPred показало высокую вероятность их взаимодействия. Анализ подтвердил полученные результаты, выявив стабильные сайты комплементарного связывания днРНК и миРНК. Построенные на этой основе сети конкурирующих эндогенных РНК показывают, что отдельные днРНК способны связывать несколько миРНК одновременно. Визуализация ключевых взаимодействий в среде Cytoscape позволила представить структуру регуляторных связей и подтвердила гипотезу о функции днРНК как “миРНК-губок” в механизмах посттранскрипционной регуляции.. Полученные результаты подтверждают, что днРНК играют важную роль в регуляторных сетях растений, действуя как модуляторы активности миРНК и влияя на экспрессию генов-мишеней. Они углубляют понимание посттранскрипционной регуляции у кукурузы и создают основу для функциональных исследований и селекционных разработок, направленных на повышение стрессоустойчивости и продуктивности растений. ПУБЛИКАЦИЯ. Янь Цзюньтин, Пронозин А.Ю., Афонников Д.А Компьютерное предсказание сети взаимодействий длинных некодирующих РНК и микроРНК кукурузы на основе транскриптома мутантной линии fuzzy tassel // Вавиловский журнал генетики и селекции. 2025. Т. 29. № 8. С. 1295-1303. doi 10.18699/vjgb-25-136