OrthoML2GO: предсказание функций белков по гомологии с использованием ортогрупп и алгоритмов машинного обучения
ВВЕДЕНИЕ. Быстрый рост объемов данных секвенирования обострил проблему функциональной аннотации белковых последовательностей, поскольку традиционные методы, основанные на гомологии, сталкиваются с ограничениями при работе с отдаленными гомологами, что затрудняет наиболее точное определение функций белков. Для преодоления ограничений, накладываемых имеющимися в настоящее время биоинформатическими методами аннотации белковых последовательностей, нами разработан новый, не имеющий аналогов метод предсказания функций белков OrthoML2GO. Фундаментальная и прикладная значимость этого метода состоит в возможности более эффективной и глубокой аннотации белковых последовательностей человека, животных, растений и микроорганизмов. ОБЩЕЕ ОПИСАНИЕ ПРОГРАММНОГО МОДУЛЯ. Программа OrthoML2GO предназначена для автоматической функциональной аннотации белковых последовательностей терминами Gene Ontology на основе поиска гомологии, анализа эволюционных отношений и машинного обучения. Программа состоит из трех блоков (рисунок Ж.44): (1) поиск гомологичных последовательностей в базе OrthoDB с использованием алгоритма USEARCH, (2) определение ортологической группы анализируемого белка методом голосования среди k-ближайших гомологов, (3) верификация предсказанных GO-терминов с помощью градиентного бустинга для исключения ложноположительных предсказаний.
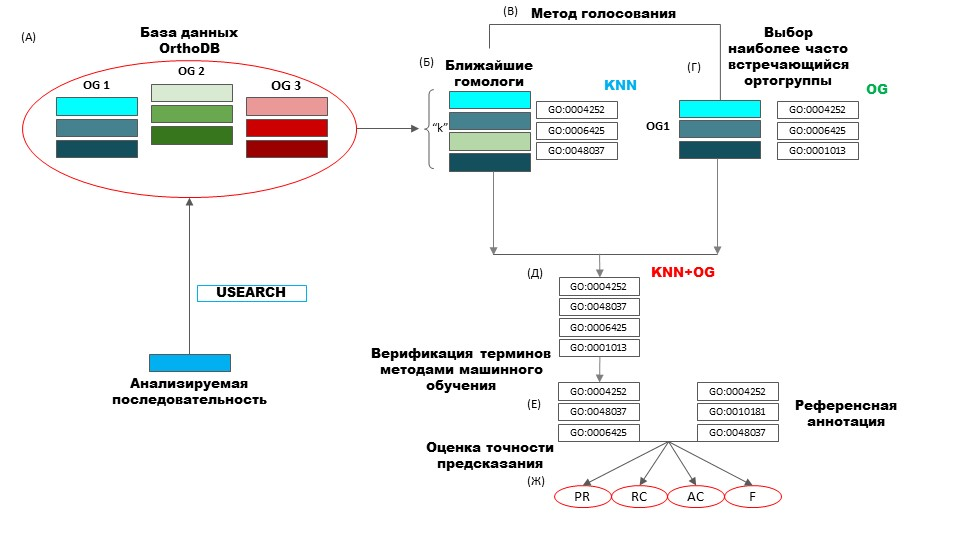
Рисунок Ж.44 – Блок схема программного модуля OrthoML2GO
Для запуска программы необходимы белковые последовательности, подлежащие аннотации, и база данных OrthoDB, содержащая информацию о гомологах, ортологических группах и известных функциях белков. Результатами работы программы OrthoML2GO являются: предсказанные GO-термины для каждой анализируемой последовательности с оценками вероятности по трем аспектам Gene Ontology (клеточные компоненты, молекулярные функции, биологические процессы), а также статистические данные о качестве аннотации (значения метрик Precision, Recall и F-score). Программа предназначена для крупномасштабной автоматической функциональной аннотации белков из геномов и протеомов различных организмов, особенно эффективна при работе с неизученными или слабоаннотированными организмами. ОПИСАНИЕ ВХОДНЫХ ДАННЫХ. На вход программы OrthoML2GO подаются белковые последовательности в формате FASTA и путь к локальной копии базы данных OrthoDB. Белковые последовательности должны содержать полные или частичные аминокислотные последовательности исследуемых белков, либо могут быть получены трансляцией из нуклеотидных последовательностей. База данных OrthoDB должна содержать информацию о гомологичных последовательностях, их ортологической классификации и известные GO-аннотации для уже аннотированных белков. ОПИСАНИЕ РАБОТЫ МОДУЛЯ И ГЕНЕРИРУЕМЫХ ВЫХОДНЫХ ДАННЫХ Программа OrthoML2GO направлена на функциональную аннотацию белковых последовательностей и состоит из трех основных этапов: · Поиск гомологичных последовательностей включает выполнение локального поиска в базе OrthoDB с использованием алгоритма USEARCH с заданными параметрами идентичности и статистической значимости, а также ранжирование найденных гомологов по степени сходства с анализируемой последовательностью. · Определение ортологической группы включает извлечение информации об ортологической принадлежности каждого из k-ближайших гомологов из базы OrthoDB, выбор наиболее часто встречающейся ортогруппы методом голосования и получение аннотации GO-терминов для выбранной ортогруппы. · Верификация и уточнение GO-терминов осуществляется с помощью градиентного бустинга, который на основе признаков сходства последовательностей и частоты встречаемости терминов исключает ложноположительные предсказания и определяет вероятность корректности каждого GO-термина. Выходными данными программы являются: списки предсказанных GO-терминов для каждой анализируемой последовательности с вероятностными оценками по трем аспектам Gene Ontology (молекулярные функции, биологические процессы, клеточные компоненты), статистические показатели качества аннотации (значения Precision, Recall, F-score и Fmax), а также сводные таблицы функционального состава анализируемых белковых наборов, позволяющие выявить характерные функциональные профили исследуемых организмов. Перечень программного обеспечения и баз данных, необходимых для работы ипм, включая блок-схему: OrthoDB v12.0, USEARCH v11.0.667, R-пакеты: xgboost, randomForest, dplyr v1.1.4. ДЕМОНСТРАЦИОННАЯ ЗАДАЧА: оценка модуля orthoml2go в сравнении с другими программными ресурсами. Проведено сравнение трёх методов предсказания функции генов. (1) Метод KNN (ближайшего соседа), использующий сходство только по гомологии. В этом случае искомой последовательности присваиваются термины GO 30 наиболее гомологичных последовательностей из базы данных OrthoDB. (2) Метод KNN+OG (ближайшего соседа с добавлением информации об аннотации ортологов). В этом случае к терминам GO, полученным методом KNN, добавляются термины, которыми аннотированы последовательности ортологической группы из БД OrthoDB, включающей наибольшее число ближайших гомологов. (3) Метод OrthoML2GO: для терминов GO оценивается вес, который зависит от уровня гомологии, аминокислотного состава и других свойств гомологичной последовательности и вычисляется с помощью метода градиентного бустинга (XGB). Этот метод продемонстрировал наилучшую точность. Было проведено его сравнение с существующими инструментами Blast2GO и PANNZER на выборках белковых последовательностей человека, дрозофилы, данио-рерио, арабидопсиса, картофеля, риса и хламидомонады. Результаты сравнения приведены таблице Ж.9. Оценивались две меры точности: F1 (среднее геометрическое между точностью и полнотой) и Fmax (максимальное значение F1, рассчитанное для всех пороговых значений предсказания функции белка). Можно видеть, что наибольший прирост точности метода OrthoML2GO достигается за счет синергии между информацией о ближайших гомологах и ортогруппах в сочетании с использованием методов машинного обучения. Полученные результаты показывают, что использование эволюционной информации, заключенной в ортогруппах OrthoDB, в сочетании с алгоритмами машинного обучения обеспечивают эффективную стратегию для автоматического предсказания функций белковых последовательностей. Предложенный метод OrthoML2GO может стать хорошей альтернативой уже существующим методам.
ПУБЛИКАЦИЯ. Малюгин Е.В., Афонников Д.А. OrthoML2GO: предсказание функций белков по гомологии с использованием ортогрупп и алгоритмов машинного обучения // Вавиловский журнал генетики и селекции. 2025. Т. 29. № 7. С. 1145-1154. doi 10.18699/vjgb-25-119